L'enzyme CP4 EPSP Synthase d'Agrobactérium tumefaciens
tolérante au glyphosate
L'enzyme CP4 5-EnolylPyruvyl-Shikimate 3-Phosphate Synthase (CP4 EPSPS) de la bactérie du sol Agrobacterium tumefaciens (souche CP4) catalyse la même réaction que la EPSPS des plantes ou de E.coli mais elle est résistante au glyphosate (classeII). C'est cette enzyme que la transgenèse fait produire au Soja OGM 40-30-2.
Mise en évidence de la tolérance de l'enzyme CP4 EPSPS au glyphosate
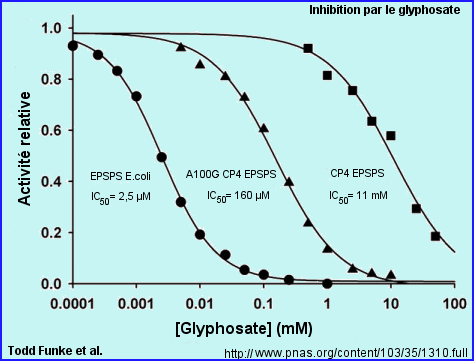
Dans l'étude proposée par T. Funke et al. l'activité de l'enzyme est déterminée par la quantité de Pi (phosphate inorganique) libéré par la réaction sur une durée de 3 min (mesure au spectro). Les 2 substrats S3P et PEP sont à concentration saturante.
IC50 correspond à la concentration en glyphosate pour laquelle l'activité enzymatique est réduite de 50%.
Les courbes montrent que l'enzyme "sauvage" de E.coli est inhibée pour des concentrations en glyphosate qui n'ont aucun effet sur l'activité de l'enzyme "sauvage" CP4 EPSPS.
IC50 CP4 EPSPS = 4400 x IC50 EPSPS.
On remarque que l'enzyme CP4EPSPS n'est tout de même pas complètement insensible au glyphosate, ce qui suggère qu'elle doit pouvoir le fixer (très faible affinité en présence de PEP).
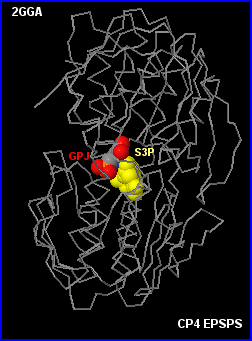
Au niveau moléculaire, la tolérance au glyphosate reste incomplètement comprise même si les études comparatives EPSPS / CP4 EPSPS incluant leurs variants (mutations dirigées) dans les domaines de la cinétique et de la biochimie structurale (analyse des modèles moléculaires) fournissent des informations intéressantes.
- Données fournies par l'étude des modèles moléculaires
Le modèle ternaire CP4 EPSPS-glyphosate (GPJ)-premier sustrat (S3P) montre que l'enzyme a une structure tertiaire très proche de celle d'E. coli (même si les 2 enzymes n'ont que 46 % d'homologie) et qu'elle peut (comme prévu) fixer le glyphosate dans le site actif à la place du deuxième substrat PEP
L'enzyme est ici en
configuration fermée (S3P fixé).
L'analyse comparative globale des modèles EPSPS et CP4 EPSPS ne fournit pas d'informations susceptibles d'expliquer les différences de sensibilité des 2 enzymes vis à vis du glyphosate, il faut approfondir l'étude en la focalisant sur l'architecture du site actif au voisinage du glyphosate.
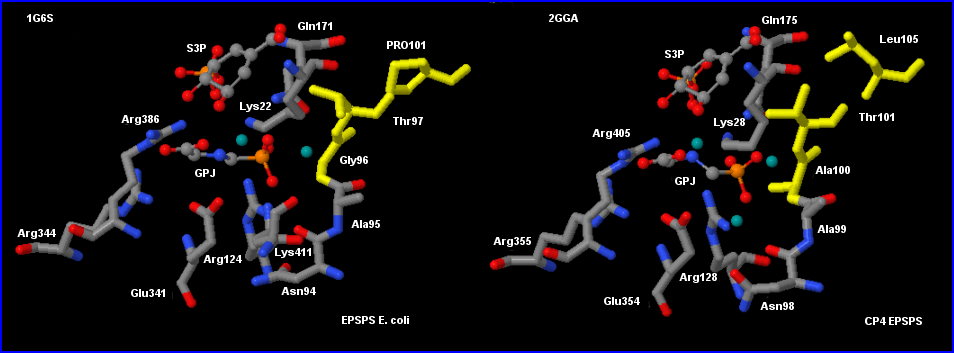
Les 2 modèles sont restreints aux résidus ac.aminés qui ont au moins une partie de leur molécule située à une distance inférieure ou égale à 0,3 nm du glyphosate. Ces ac. aminés sont susceptibles d'établir, par certains atomes, des liaisons hydrogène avec le glyphosate, soit directement soit par l'intermédiaire des molécules d'eau présentes (vert-gris). Il y a en plus Pro101 sur le modèle à gauche et Leu105 sur celui de droite qui sont situés plus loin (0,9 nm).
La plupart des ac.aminés impliqués dans la détermination de la configuration spatiale de la partie du site actif qui accueille le glyphosate (et PEP) se retrouvent dans les 2 enzymes. Gly 96 est remplacé par Ala100 dans la synthase CP4EPSP. Le groupement -CH3 de Ala100 est orienté vers l'intérieur de la cavité où se place le glyphosate et en diminue le volume par encombrement stérique: le glyphosate a dans le complexe qu'il forme avec CP4EPSPS et S3P une configuration un peu plus "ramassée" que dans le complexe correspondant qu'il forme avec EPSPS (configuration étalée).
Lys401, résidu du site actif de EPSPS E.coli, n'a pas d'équivalent dans celui de CP4EPSPS.
Rq/ Le remplacement de Pro101 par Leu 105 ne concerne pas directement le site actif mais a des conséquences sur l'orientation de Thr97 / Thr101 ce qui peut influer sur la configuration du site d'accueil du glyphosate (et du PEP).
- Données fournies par l'analyse des cinétiques enzymatiques
Les études cinétiques réalisées avec le variant Ala100Gly (obtenu par mutagenèse dirigée) de CP4EPSPS (voir courbes ci-dessus) montre une augmentation très significative de la sensibilité au glyphosate de l'enzyme modifiée. Inversement, le variant de EPSPS E.coli, Gly96Ala est plus tolérant vis à vis du glyphosate que l'enzyme "sauvage" (son affinité pour PEP diminue).
La substitution Pro101 / Leu 105 entraîne une baisse significative de la sensibilité au glyphosate pour l'enzyme EPSPS de E. coli.
- Synthèse
- Grande sensibilité au glyphosate des EPSPS des végétaux et de certaines bactéries (dont E. coli)
Le glyphosate est un inhibiteur de l'enzyme qui entre en compétition avec le PEP (2ème substrat) alors que le 1er substrat, S3P, est déjà en place dans le site actif. Quand le glyphosate est employé comme principe actif dans un herbicide, les conditions (concentration) sont telles que l'enzyme fixe préférentiellement le glyphosate dans sa configuration étalée. Le complexe EPSPS-S3P-glyphosate a alors une très grande probabilité de se former aux dépens du complexe EPSPS-S3P-PEP. Quand le glyphosate est en place, il occupe le site qui accueille normalement le PEP, les 2 substrats ne peuvent pas cohabiter dans le site actif de l'enzyme, la réaction ne se produit pas.
Ainsi, la vitesse de la réaction directement dépendante de la quantité de complexes EPSPS-S3P-PEP formés par unité de temps est très faible ou nulle en présence de l'inhibiteur glyphosate.
- Tolérance de CP4 EPSPS (Agrobacterium tumefaciens) au glyphosate
Les enzymes CP4 EPSPS et EPSPS ont un site actif avec une configuration très proche (la plupart des ac. aminés sont conservés). CP4 EPSPS accueille les substrats S3P et PEP avec une affinité comparable à celle de EPSPS mais ne peut fixer le glyphosate que dans une conformation "ramassée" qui confère au complexe CP4 EPSPE-S3P-glyphosate une certaine instabilité le rendant donc (énergiquement) beaucourp moins probable que le complexe CP4 EPSPS-S3P- PEP.
Une simple substitution comme EPSPS Gly96 / CP4 EPSPS Ala100 peut expliquer la différence d'affinité pour le glyphosate entre les 2 enzymes, Ala entraînant par la présence de son -CH3 ,un encombrement stérique qui contraint le glyphosate à prendre la conformation "ramassée" mais n'enpêche pas le PEP (plus petit) de prendre place dans le site actif. D'autres résidus ac. aminés peuvent également être impliqués.
Ainsi lorsque le glyphosate d'un herbicide est mis en présence de l'enzyme CP4 EPSPS, il ne se forme pratiquement que des complexes CP4 EPSPS-S3P-PEP( compétition en faveur du 2ème substrat) , la réaction catalysée par l'enzyme a lieu.