
Un test pour révéler la présence d'OGM
dans
l'alimentation humaine et animale
Pour caractériser la présence d'OGM dans un aliment, le test peut soit cibler la cassette d'expression du transgène (ADN) soit la protéine codée par ce transgène.
Le test qui vise la protéine d'intérêt, CP4EPSPS pour le Soja, est de type ELISA, il est basé sur l'utilisation d'anticorps spécifiques de cette protéine. Il n'est pas décrit ici.
Les tests qui révèlent la présence (ou l'absence) de la cassette d'expression du gène d'intérêt sont soit spécifiques soit à spectre large, ils sont basés sur l'amplification par PCR de séquences sélectionnées dans la cassette. Certains pemettent de quantifier la teneur en OGM (PCR en temps réel), d'autres ne sont que qualitatifs. Pour ces derniers, certains sont spécifiques d'un OGM donné car ils ciblent au moins une partie de la séquence codante du transgène alors que d'autres sont "généralistes" car ils cherchent à mettre en évidence des séquences incluses dans le promoteur P-E35S et dans le terminateur NOS3' très souvent employés pour la transgenèse végétale (environ 80% des OGM commercialisés). C'est ce dernier test qui sera décrit ici car c'est celui qui est utilisé en TP, avec le kit Bio-Rad.
Principe du test "généraliste" basé sur la PCR
- Extraire l'ADN de l'aliment. Eviter les contaminations.
- Vérifier si de l'ADN végétal en bon état a bien été extrait : rechercher par PCR le gène qui code une protéine du photosystème II, présent chez la plupart des végétaux (et bien conservé) ou le gène de la lectine du Soja (si la recherche est spécifique).
- Rechercher par PCR, dans l'ADN extrait, la présence (ou l'absence) du promoteur P-E35S et du terminateur NOS3'.
La séparation et l'identification des produits de PCR sont réalisées par électrophorèse en gel d'agarose contre un marqueur de taille.
Le protocole n'est pas décrit ici en détails car les élèves le découvrent en le mettant en oeuvre au cours de la séance de TP.
Rq/ Principe de la PCR : voir site SVT Lycée.
Il semble intéressant, pour mieux appréhender l'intervention de la PCR dans le protocole, de proposer aux élèves un travail de recherche (logiciel Primer3) qui consiste à prévoir la taille des amplicons qui caractérisent respectivement le promoteur et le terminateur, en connaissant la séquence des amorces utilisées.
Prévision de la taille des amplicons caractéristiques du promoteur E-P35S et du terminateur NOS3'
Cette prévision ne peut malheureusement pas être réalisée pour les fragments amplifiés en TP pour détecter la présence des 2 séquences car nous utilisons le kit Bio-Rad et le fournisseur refuse de communiquer la séquence des amorces qu'il commercialise.
Les amorces proposées ici sont très souvent utilisées par les labos officiels qui cherchent à détecter la présence d'OGM dans les aliments.
-couple d'amorces (p35s-cf3 et p35s-cr4) pour le promoteur P-E35S:
left 5'CCACGTCTTCAAAGCAAGTGG3'; right 5'TCCTCTCCAAATGAAATGAACTTCC3'
-couple d'amorces (HA-nos118-f et HA-nos118-r) pour le terminateur NOS3'
left 5'GCATGACGTTATTTATGAGATGGG3'; right 5'GACACCGCGCGATAATTTATCC3'
Affichage graphique du positionnement des amorces et des amplicons avec Primer Blast
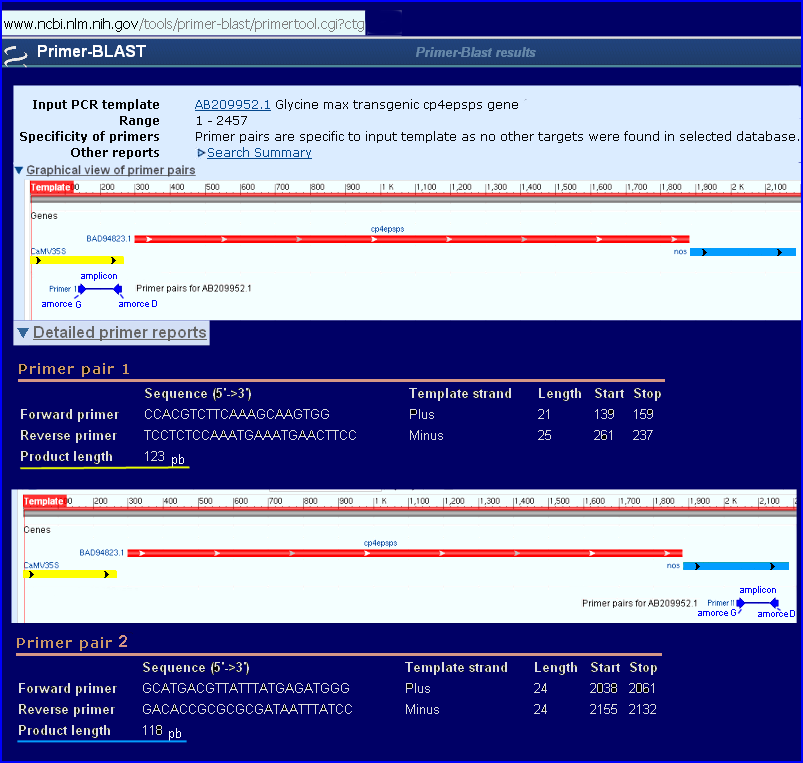
Positionnement des amorces dans la séquence avec Primer3
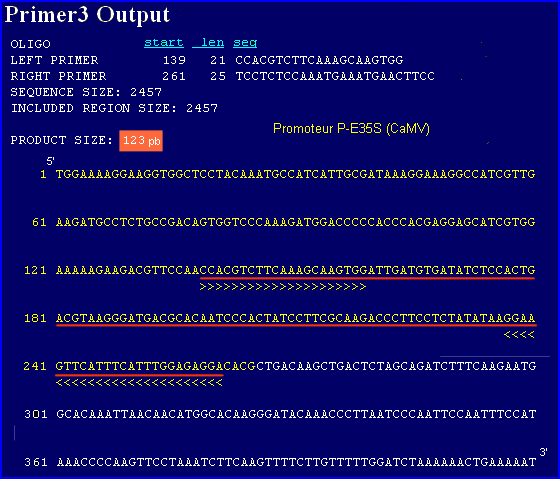
Couple d'amorces : p35s-cf3 et p35s-cr4
Taille prévue pour l'amplicon qui caractérise la présence du promoteur P-E35S (CaMV35S) : 123 pb
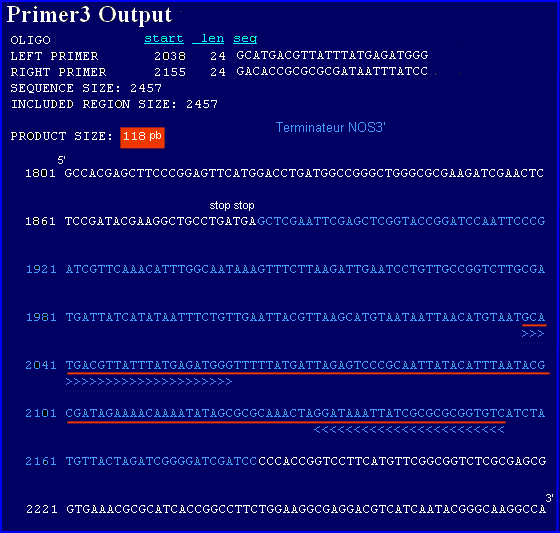
Taille prévue pour l'amplicon qui caractérise la présence du terminateur NOS3': 118 pb
Résultat d'un test PCR réalisés avec les couples d'amorces p35S-cf3 / p35s-cr4 et HA-nos118-f / HA-nos118-r

Amorces p35S-cf3 et p35s-cr4
Détection du promoteur P-E35S
-1,2,3 : graines de Soja crues
- 4 : poudre de Soja référence
(
avec % Soja 40-3-2 GM connu)
- 5 : ADN contrôle -
-MT : marqueur de taille
Détection du terminateur NOS3'
- 1,2,3 : graines de Soja crues
- 4 : ADN contrôle +
- 5 : ADN contrôle -
- MT : marqueur de taille