De la collecte des cellules
au profil ADN multi-loci (STRs)
Comment procède-t-on pour obtenir puis analyser des empreintes génétiques dans le cadre d'une enquête criminelle?
Collecte des cellules qui fournissent l'ADN
Quelques cellules peuvent être récupérées à partir de traces (sang, sperme, salive...) ou d'un cheveu ou encore d'un fragment de peau... laissés sur les lieux du crime. Un prélèvement de cellules est également réalisé sur la victime et sur les suspects (sang ou cellules de l'épithélium buccal).
Les prélèvements doivent être réalisés avec méthode en évitant toute contamination.
Extraction de l'ADN
Voir Tp pv92 site SVT lycée
Amplification des marqueurs par PCR multiplex
De très faibles quantités d'ADN (de l'ordre du ng) extrait des cellules collectées peuvent suffire. On amplifie, en une seule réaction (dans un même tube) plusieurs marqueurs. Il faut utiliser un couple d'amorces par marqueur. Les STRs ciblés ont en général un motif répété de 4 nucléotides (comme TH01), ils se prêtent bien à une amplication par PCR.
La technologie utilisée pour identifier les fragments d'ADN amplifiés nécessite leur "marquage" par un fluorophore qui est porté par les amorces. Pour un marqueur donné, seule une amorce est marquée par son extrémité 5'.
Séparation par électrophorèse capillaire des fragments d'ADN amplifiés
Les allèles d'un marqueur STRs ne diffèrent que de 4, 8, 12.... nucléotides,
suivant le nombre de motifs (4 nucléotides) répétés. Pour certains (ex allèles 9.3 et 10 de TH01), la différence de taille ne porte que sur un nucléotide. Pour séparer les fragments d'ADN amplifiés (amplicons), correspondant aux allèles des différents marqueurs, on utilise un système qui permet une électrophorèse à très haute résolution , c'est l'électrophorèse capillaire.
L'échantillon contenant les produits amplifiés (après PCR) est injecté (voir ci-dessous) dans un tube capillaire long (47 cm) de très faible diamètre interne (50 µm) rempli d'un gel de faible viscosité (polymère). Un très forte tension (15 kV) est appliquée au contenu du capillaire maintenu à température constante. Les fragments d'ADN dénaturé (monobrin) chargés négativement, soumis au champ électrique, migrent dans le gel, vers l'anode d'autant plus rapidemment qu'ils sont plus courts.
Détection par fluorescence des fragments d'ADN séparés par électrophorèse capillaire
A l'issue de la PCR, les amplicons des différents STRs se retrouvent marqués par le fluorophore couplé à l'une des amorces du couple qui a permis leur amplification. Il est important de pouvoir associer des fluorophores différents à des marqueurs susceptibles de fournir des fragments de taille très proche.
Une fenêtre de détection est disposée le long du capillaire. Quand les fragments d'ADN parviennent à son niveau, plus ou moins séparés les uns des autres après avoir migré sur une distance suffisante (par exemple 36 cm), le fluorophore qu'ils portent est excité par un laser. Il se désexcite en émettant des photons par fluorescence.
Une collection de marqueurs "standard de taille" (avec un fluorophore particulier) est ajoutée à chaque tube à tester et traitée avec l'échantillon. Un mélange des principaux allèles des STRS analysés (allelic ladder) est régulièrement injecté dans le capillaire et traité comme un échantillon, les allèles d'un marqueur donné étant marqués comme ceux qui ont été amplifiés et qui se retrouvent dans un échantillon.
Traitement des données de fluorescence
Les signaux fluorescents subissent une décomposition spectrale (prisme) et chaque faisceau est dirigé vers une caméra CCD qui transforme le signal lumineux en un signal électrique converti en information numérique utilisable par les logiciels.
Finalement, quand les nombreux fragments (amplicons) correspondant à un allèle d'un marqueur passent groupés au niveau de la fenêtre de détection, il y a une émission fluorescente qui se traduit en fin de traitement par un pic caractérisé par sa position (donc par la taille de l'amplicon) et par l'intensité de la fluorescence exprimée en Unité de Fluorescence Relative.
Les pics de la collection d'allèles de référence (allelic ladder) peuvent être résolus en taille relativement aux marqueurs standards. Les pics des échantillons à analyser peuvent être convertis en allèles d'un STR donné, affichés en nombre de motifs répétés, par comparaison avec les pics des allèles de référence.
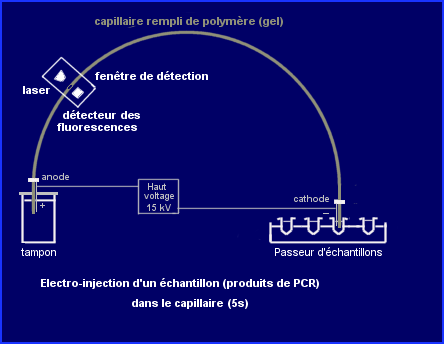
Pour la configuration "électrophorèse / détection", survolez l'image avec la souris.
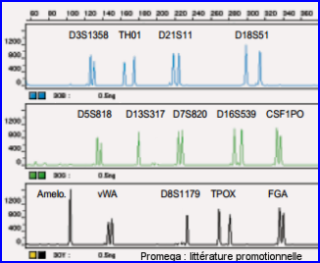
analyse de 13 STRs + un marqueur du sexe (Amelo)